L’impronta genetica della biodiversità lucana
Nel centro ricerche dell'ALSIA "Metapontum Agrobios" individuati marcatori molecolari delle specie agrarie, indipendenti dallo stadio fenologico della pianta o da variabili ambientali

Apparecchiature utilizzate dal centro ricerche Metaponum Agrobios di ALSIA per il sequenziamento di nuova generazione (NGS).
Data:Thu Oct 29 16:39:35 CET 2020
L’Agenzia Lucana di Sviluppo ed Innovazione in Agricoltura (ALSIA), attraverso la rete delle sue aziende sperimentali, è da anni impegnata nel recupero, moltiplicazione, catalogazione e conservazione di germoplasma tipico della Basilicata. Le risorse genetiche di interesse agrario, oltre che conservate e custodite presso le aziende degli agricoltori custodi e nei campi catalogo delle aziende sperimentali dimostrative di ALSIA, sono sottoposte ad attività di caratterizzazione fenotipica e a prove pluriennali di valutazione agronomica che hanno come risultato, molto spesso, la selezione di nuovi ecotipi di interesse per il territorio e lo sviluppo di filiere di prodotti tipici lucani.
Alla valorizzazione delle risorse genetiche vegetali l’ALSIA contribuisce anche con il suo Centro Ricerche "Metapontum Agrobios". Il centro ricerche, con le competenze professionali e le strumentazioni all’avanguardia, è infatti impegnato nella caratterizzazione del germoplasma lucano a livello del DNA con analisi che consentono di individuare tratti distintivi (impronta genetica) delle diverse specie agrarie.
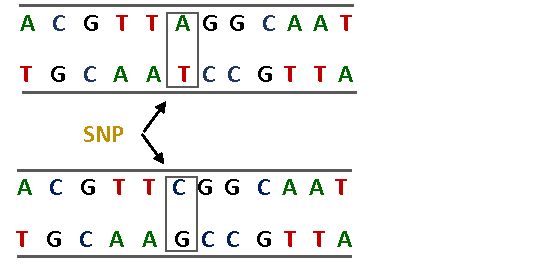
La variabilità delle sequenze geniche è fondamentale per le analisi di caratterizzazione genetica e l'ALSIA, attraverso lo studio del DNA, individua le differenze (variabilità) presenti a livello di singolo nucleotide, tecnicamente chiamate SNP (Single Nucleotide Polymorphisms, Figura 1), molto comuni e abbondanti nel genoma di tutti gli esseri viventi. Le differenze nucleotidiche consentono di differenziare varietà ed ecotipi, o addirittura individui all’interno di una stessa specie.
Figura 1. Esempio di differenza nucleotidica singola (SNP). Nella figura sono schematizzati due frammenti di DNA che differiscono, nella stessa posizione, per un singolo nucleotide circoscritto dai rettangoli ed indicato dalle frecce.
 |
Le tecnologie e le metodologie oggi a disposizione per il rilievo della variabilità nel DNA sono tante e diverse. Presso il centro di ricerca di ALSIA gli SNP vengono identificati mediante una metodologia (RESTseq, RESTriction fragment SEQuencing) che si basa sul sequenziamento di nuova generazione (NGS, Next Generation Sequencing). La tecnologia permette di generare contemporaneamente, in breve tempo ed a basso costo, milioni di sequenze geniche e di informazioni.
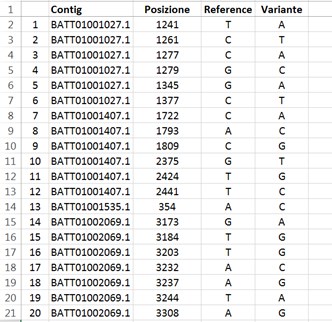
I dati prodotti dal sequenziamento, confrontati con le informazioni presenti nelle banche dati pubbliche relative ai genomi delle specie già sequenziate (reference), consentono di individuare le differenze nucleotidiche. Si genera così un profilo genetico molto dettagliato, caratterizzato da migliaia di SNP ciascuno dei quali ben identificato per la sua la posizione nel genoma della specie oggetto dell’analisi (Figura 2).
Figura 2. Esempio di profilo genetico generato dal confronto dei dati del sequenziamento con le informazioni presenti in banca dati (Reference). Per ogni SNP (Variante) identificato è riportata la sua posizione all’interno della molecola del DNA (Contig).
 |
Sono diversi gli studi in corso sul profilo genetico di genotipi e varietà tradizionali coltivate in Basilicata.
La metodologia RESTseq, è stata utilizzata dall'ALSIA per la caratterizzazione genetica di un panel di 29 ecotipi di fagiolo provenienti da due aree distinte della Basilicata: l’area del Pollino (19 accessioni della banca del germoplasma di ALSIA) e quella della Val d’Agri (10 accessioni di fagiolo di Sarconi). Mediante il sequenziamento di nuova generazione sono stati identificati 3.405 SNP nel panel di fagiolo. L’informazione prodotta dalla posizione e identità delle differenze nucleotidiche (SNP) ha permesso di generare una distinta “impronta genetica” per ciascuna delle accessioni di Phaseolus vulgaris analizzata.
Tra i 3.405 SNPs sono stati individuati alcuni SNP che possono essere utilizzati come marcatori molecolari nelle analisi di identificazione varietale. Per esempio nelle due DOP Fagiolo Poverello e Fagiolo Bianco di Rotonda sono stati individuati 3 SNP che possono essere impiegati nel riconoscimento varietale o per il controllo della stabilità genetica dei due ecotipi o per la loro tutela e protezione da frodi. Tali SNP, localizzati su 3 diversi cromosomi del genoma di Phaseolus vulgaris, sono stati validati da altri approcci molecolari e testati con successo su una popolazione più ampia dei due ecotipi di fagiolo.
Anche la melanzana rossa di Rotonda (Figura 3), altro prodotto a marchio (DOP) della Basilicata, è stata caratterizzata molecolarmente. Dall’analisi del suo genoma sono stati identificati ben 3.216 SNP che hanno permesso di stabilire ad esempio che la melanzana DOP della valle del Mercure, visivamente indistinguibile da quella rossa di Maratea, è identica geneticamente a quest’ultima.
Figura 3. Melanzana rossa di Rotonda
 |
Un profilo genetico molto dettagliato è stato generato anche per la Carosella, una delle più antiche popolazioni di frumento esaploide dell’area del Pollino. E’ noto che la Carosella è un miscuglio e al suo interno, sulla base di alcuni tratti del fenotipo della spiga, del colore del glume e della presenza/assenza delle reste, sono state individuate sei sottopopolazioni.
L’identificazione di 2.841 SNP (differenze nucleotidiche) nel genoma dei 6 sottogruppi fenotipici della Carosella e il loro impiego in analisi di distanza genetica hanno rivelato che le sottopopolazioni della Carosella, fenotipicamente distinte in numero di 6, dal punto di vista genetico sono invece raggruppabili in 5 sottopopolazioni.
Nel progetto FiNoPOM (acronimo di Fico, Nocciolo e Pomacee) della Sottomisura 10.2 del PSR Basilicata 2014-2020, il centro ricerche di ALSIA è invece impegnato nella caratterizzazione genetica di antiche varietà di alberi da frutto e precisamente di fico, nocciolo, pero e melo (117 in totale) censite e recuperate nell’area del Pollino. Molte delle varietà sono già iscritte nel Repertorio regionale della Biodiversità (art. 3 LR 26/2008), altre ancora da iscrivere.
Dall’analisi e confronto dei profili genetici prodotti per diverse varietà di fruttiferi sono stati individuati una serie di SNP genotipo-specifico molto promettenti ad essere utilizzati come marcatori molecolari nelle analisi di identificazione varietale. Tali marcatori potrebbero essere impiegati, ad esempio, per il censimento e riconoscimento varietale del germoplasma presente su tutto il territorio lucano o come supporto per risolvere casi di sinonimia (stesso genotipo con nomi diversi).
La disponibilità di marcatori molecolari per le specie agrarie di interesse è molto utile e vantaggiosa rispetto alle altre tipologie di marcatori (per es. morfologici o biochimici) in quanto i tratti distintivi (SNP) di ciascun genotipo, poiché intrinsechi nel DNA, possono essere utilizzati in qualsiasi momento per il monitoraggio dell’identità delle accessioni. Tali marcatori sono infatti svincolati dalle stagioni o dallo stadio fenologico della pianta e, soprattutto, non sono influenzati dalle variabili ambientali.
